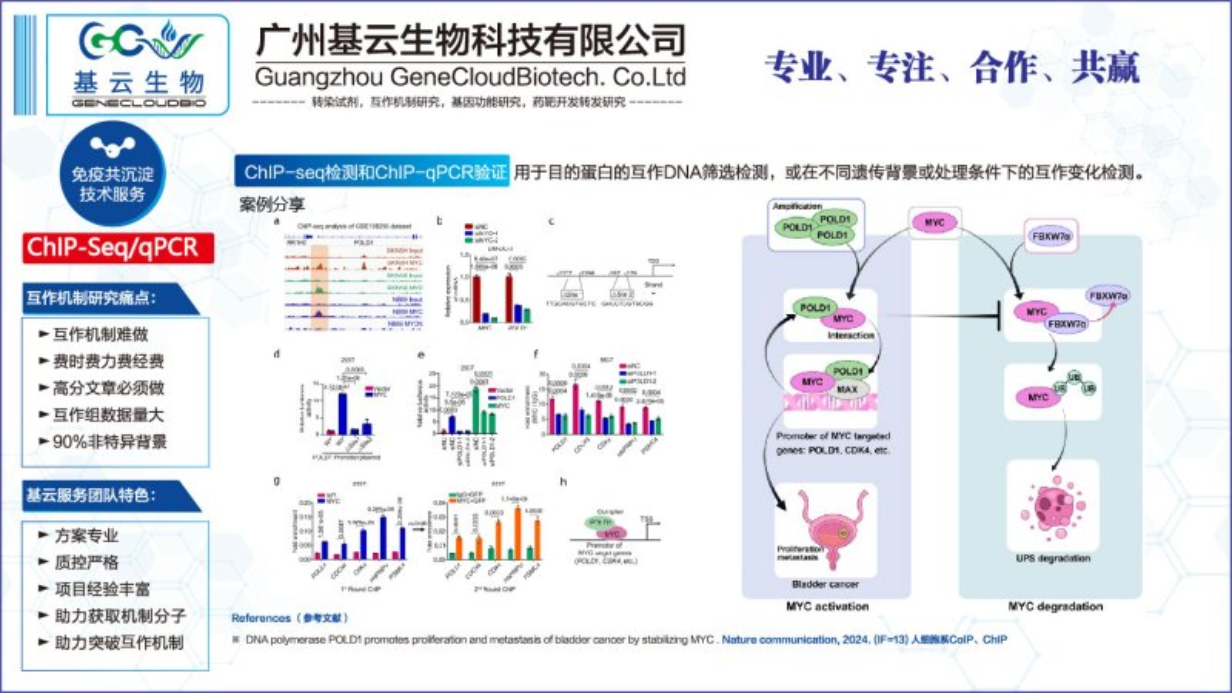
ChIP-Seq检测原理:ChIP-Seq检测原理和RIP-Seq类似,不同的是前者利用目的蛋白抗体将相应的DNA-蛋白复合物沉淀下来,然后分离纯化捕获DNA,结合高通量测序技术对目标DNA进行测序分析。ChIP-Seq服务要点和RIP-Seq类似,精简如下:(1)试验设计:同RIP-Seq。(2)蛋白表达和细胞量:比RIP-Seq细胞用量要求大,建议不少于10e7(金标准:320g离心沉淀100ul)。(3)抗体关键质控:同IP-Mass和RIP-Seq。(4)IP送样建议:细胞培养好后,收集前,先进行交联,再收样冻存。(5)互作DNA筛选和验证:同RIP-Seq。ChIP-Seq优劣势:优势:高通量获得目的蛋白的专属DNA互作库。劣势:技术门槛高,一般需要整包交给专业的服务商开展检测。ChIP-Seq应用扩展:(1)蛋白DNA相互作用数据,是探究转录调控机制研究的重要内容,体现机制研究的深度,能显著提高临床基础类研究文章的档次。(2)蛋白DNA互作组检测,常用于蛋白的转录调控研究,如转录因子,转录调控蛋白等。(3)蛋白DNA互作,其结合DNA的区域,是进一步研究互作机制和功能的关键内容,能够显著提高机制研究的高度。为什么要进行ChIP-qpcr实验。云南chromatin蛋白互作ChIP
ChIP-seq与ChIP-qPCR在实验技术、分辨率和数据分析方面存在明显的不同之处。首先,ChIP-seq结合了高通量测序技术,能够在全基因组范围内检测蛋白质与DNA的结合位点。它通过测序仪对富集的DNA片段进行大规模并行测序,生成海量的数据,从而提供高分辨率的结合位点信息。相比之下,ChIP-qPCR则侧重于对特定基因或基因区域进行定量分析,它通过荧光定量PCR技术检测富集的DNA片段的数量,具有更高的灵敏度和特异性,但只能针对已知序列进行分析。其次,ChIP-seq在分辨率上优于ChIP-qPCR。由于ChIP-seq可以对全基因组进行测序,它能够检测到更多的结合位点,包括那些低丰度或远离转录起始位点的结合事件。而ChIP-qPCR则受限于所选择的基因或基因区域,可能无法全局反映蛋白质在基因组上的结合情况。在数据分析方面,ChIP-seq生成的数据需要进行复杂的生物信息学分析,包括序列比对、峰值调用、注释和富集分析等步骤。而ChIP-qPCR的数据分析相对简单,主要通过比较不同样品间的荧光信号强度来判断蛋白质的结合情况。云南chromatin蛋白互作ChIPChIP-seq实验技术基本原理是什么。

在考虑进行ChIP-qPCR实验时,通常涉及以下情况:验证特定蛋白质与DNA的结合:当你有明确的假设,认为某个特定的转录因子、组蛋白或其他染色质相关蛋白质与某个基因或基因区域结合时,ChIP-qPCR是一种有效的验证方法。定量分析蛋白质与DNA的结合程度:ChIP-qPCR允许你对特定基因或基因区域的蛋白质结合进行定量分析,这对于比较不同条件下(如不同时间点、不同处理或不同细胞类型)的结合差异特别有用。研究少量基因或特定区域:与ChIP-seq相比,ChIP-qPCR更适用于研究少量基因或特定基因区域,因为它更经济、更快速,并且对于特定目标的检测具有更高的灵敏度。资源有限:当你没有足够的资源或时间进行全基因组的ChIP-seq分析时,ChIP-qPCR可以作为一个更可行且成本效益更高的选择。初步筛选或验证:在进行更大规模的ChIP-seq实验之前,ChIP-qPCR可以作为初步筛选或验证特定蛋白质与DNA结合位点的有效工具。在这些情况下,ChIP-qPCR提供了一种灵活、敏感且经济高效的方法来研究蛋白质与DNA的相互作用。
染色质免疫沉淀(ChIP)实验的优点(一)。高特异性:ChIP技术可以针对特定的染色质修饰或蛋白进行检测,具有很高的特异性。通过使用特异性抗体,可以精确地识别并沉淀与目的蛋白结合的染色质片段,从而研究该蛋白在基因组上的结合位点。保存染色质结构:ChIP实验可以在细胞或组织中保留染色质的原始状态,包括其三维结构和局部环境。这有助于研究蛋白质与染色质之间的相互作用以及染色质的结构和功能。可定量分析:ChIP技术可以定量测定染色质修饰或蛋白-DNA结合的丰度,从而提供定量的分析结果。通过比较不同样品或条件下的ChIP信号强度,可以评估蛋白质与DNA结合的相对亲和力或活性。ChIP-qPCR和ChIP-seq在实验流程、分辨率和应用范围上存在异同点,应根据具体需求选择合适的技术方法。
ChIP-qPCR和ChIP-seq实验在多个方面存在异同点。首先,在实验流程上,两者都包含染色质免疫沉淀这一关键步骤,用于富集与特定蛋白质结合的DNA片段。然而,在后续的检测方法上,它们有所不同。ChIP-qPCR采用实时荧光定量PCR技术对这些片段进行定量检测,适用于已知蛋白质与靶序列相互作用的研究。而ChIP-seq则结合了高通量测序技术,能够在全基因组范围内检测与特定蛋白质结合的DNA区域,适用于未知靶序列的探索。其次,在分辨率上,ChIP-seq具有更高的分辨率,能够提供完整、高分辨率的结合信息,绘制出转录因子等蛋白质在全基因组范围内的结合位点图谱。而ChIP-qPCR的分辨率相对较低,通常只能针对已知基因或基因区域进行分析。另外,在应用范围上,ChIP-seq在探索转录调控网络、表观遗传机制等领域具有更广泛的应用价值。而ChIP-qPCR则更适用于验证特定转录因子与基因启动子的结合等具体作用机制的研究。综上所述,ChIP-qPCR和ChIP-seq在实验流程、分辨率和应用范围上存在异同点,研究者应根据具体需求选择合适的技术方法。ChIP-seq实验流程包括细胞处理、染色质免疫沉淀、文库构建和高通量测序等步骤。染色体蛋白互作检测ChIP-RT-PCR
ChIP实验常见的应用场景有哪些。云南chromatin蛋白互作ChIP
ChIP的一般流程:甲醛处理细胞---收集细胞,超声破碎---加入目的蛋白的抗体,与靶蛋白-DNA复合物相互结合---加入ProteinA,结合抗体-靶蛋白-DNA复合物,并沉淀---对沉淀下来的复合物进行清洗,除去一些非特异性结合,洗脱,得到富集的靶蛋白-DNA复合物,解交联,纯化富集的DNA,PCR分析。在PCR分析这一块,比较传统的做法是半定量-PCR。但是现在随着荧光定量PCR的普及,大家也越来越倾向于Q-PCR了。此外还有一些由ChIP衍生出来的方法。例如RIP(其实就是用ChIP的方法研究细胞内蛋白与RNA的相互结合,具体方法和ChIP差不多,只是实验过程中要注意防止RNase,分析的时候需要先将RNA逆转录成为cDNA);还有ChIP-chip(其实就是ChIP富集得到的DNA,拿去做芯片分析,做法在ChIP的基础上有所改变,不同的公司有不同的做法,要根据公司的要求来准备样品)。云南chromatin蛋白互作ChIP